使用全基因组测序调查结核病的暴发
2013-02-22 12:22:17 来源:37度医学网 作者: 评论:0 点击:
背景
在过去的十年间,英国的结核病发病率有所上升。 疾病控制依赖于流行病学数据,而这些数据可能难以获得。 全基因组测序可以检出结核分枝杆菌菌株中的微进化。 我们目的是估算英国米德兰兹郡的相关结核分枝杆菌菌株的遗传多样性,并探讨如何将这一测量结果用于调查社区爆发。

方法
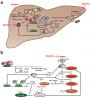
在一项回顾性观察研究中,我们使用 Illumina 公司的技术对来自存档冻存培养物的结核分枝杆菌基因组进行了测序。 我们根据菌株特点将其分为四组:横断面、纵向、家庭和社区。 我们测定了家庭暴发中宿主内和宿主间配对的核苷酸差异,并估算了 DNA 序列中的变化率。 我们根据分枝杆菌散在重复单位—可变数目串联重复序列数据建立了 11 个社区集群,并使用这些调查结果解释了根据上述集群构建的网络图。
结果
我们对来自 254 名患者的 390 个隔离群进行了测序,其中包括所有五个主要结核分枝杆菌谱系的代表。 在来自 30 名个人和 25 个家族的纵向隔离群中,估算 DNA 序列变化率为每年每基因组 0·5 个单核苷酸多态性 (SNP) (95% CI 0·3—0·7)。 差异很少高于 3 年五个 SNP。 在来自个人和家族的配对的隔离群中有 109/114 (96%) 差异不超过五个 SNP。 使用五个以上的 SNP 对来自下列患者的隔离群进行区分:69 名流行病学相关联的患者中无差异;在 13 名可能存在关联的患者中,有两人 (15%) 与其他人存在差异;75 名流行病学无关患者中有 13 人 (17%) 与其他人存在差异(三种方式对比准确性 p<0·0001)。 进化树及临床和流行病学数据显示,两个社区集群中存在超级传染者。
结果解读
全基因组测序可阐明结核病的爆发,并可用于推论病例之间的传播方向。 这项技术可识别超级传染者并预测未确诊病例的存在,从而可能对感染患者及其接触者进行早期治疗。
Walker TM, Ip CLC, Harrell RH, et al. Whole-genome sequencing to delineateMycobacterium tuberculosis outbreaks: a retrospective observational study. Lancet Infect Dis. 2013;13:137-46.
上一篇:两种股骨假体远端设计及应用特点
下一篇:2012肾脏病学之进展(一)
论坛新帖
频道总排行
医学推广
频道本月排行
热门购物
评论排行
- 2011年临床执业医师考试实践技能真...(13)
- 腋臭手术视频(11)
- 2008年考研英语真题及参考答案(5)
- 节食挑食最伤女人的免疫系统(5)
- 核辐射的定义和单位(5)
- CKD患者Tm与IMT相关(5)
- 齐鲁医院普外科开展“喉返神经监护...(5)
- windows7激活工具WIN7 Activation v1.7(5)
- 正常微循环(5)
- 美大学性教育课来真的 男女上阵亲...(4)